什么是核苷酸多样性,它为什么十分重要?
什么是核苷酸多态性?
核苷酸多态性是指测序反应每个cycle中A、T、G、C四种碱基的相对比例。平衡性或者多态性高的文库在测序反应的每个cycle中四种碱基的比例大致相当。多态性低的文库则在同一个cycle中某些核苷酸比例很高,其它核苷酸比例很低。对于某些多态性高的文库,如果带有Barcode或者其它分子标签,那么它的某些区域的多态性会比较低。
下面是MiSeq™测序的一个Cycle中一个Tile在A、T、C、G四个通道的照相Thumbnail图片,您可以看到多态性高的文库和多态性低的两种文库四种核苷酸的相对比例。如图所示,平衡文库(Well-balanced)在每个通道簇的信号密度比较接近;多态性低的文库(Low diversity),C通道簇的信号密度比较高,A,G和T通道簇的信号密度则低很多。如果所有簇的信号集中在某一个通道,会影响仪器对簇进行定位以及碱基读取的质量。

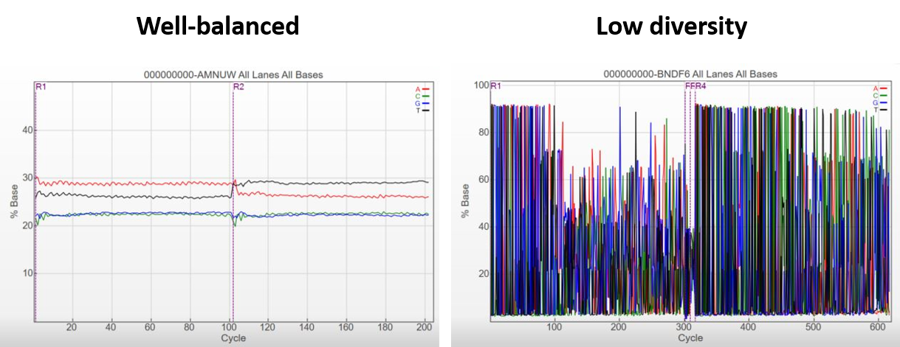
下面的图表举例说明了平衡性较好的文库和多态性低的文库测序结果用Sequencing Analysis Viewer (SAV)软件分析出的每个cycle的碱基百分比。对于平衡性好的文库(Well-balanced),四个通道的信号都占整体的25%左右。对于多态性低的文库(Low diversity),不同的cycle,每种核苷酸的相对比例会有较大的差异。某些cycle中,几乎90%的信号来自同一个通道。
为什么碱基多样性很重要?
碱基多样性对于测序质量以及高质量数据的获取至关重要。前25个Cycle又是重中之重,因为Cluster Passing Filter、phasing/pre-phasing以及Color matrix correction等参数的计算都是基于前25个Cycle的结果完成的。Phasing/Pre-phasing是一个簇中相对于当前cycle,落后(phasing)或者超前(pre-phasing)的分子比例。Color matrix correction对应于前几个cycle的模板生成,这个模板包含了每个通道的光强,会用于后续碱基读取以及phasing/pre-phasing的计算。上述这些指标都会用于后续碱基读取以及质量值的计算。如果荧光信号比较均衡,后续的模型会更准确,数据质量也会有所提高。
对于非Patterned Flow Cell,簇的数量和位置是通过前面4-7个Cycle来决定的,这个过程称为模板生成(Template Generation)。对于采用非Patterned Flow Cell的平台,包括MiniSeq™, MiSeq™, NextSeq™ 500/550, and HiSeq™ 1000/2500,模板生成过程中的碱基平衡性非常重要。四种碱基的信号在前4-7个cycle中都出现才能生成比较好的模板。
Illumina测序平台采用三种类型的SBS技术
HiSeq™和MiSeq™系统采用四通道化学。四种碱基的发射光波长都不同,每个cycle会生成四张图片。
RTA(Real-Time Analysis)软件会根据经验值来计算color normalization matrix以及phasing/pre-phasing的值。二者都会用于后续的碱基读取和质量值的计算。MiSeq和HiSeq2500使用的是RTA1,HiSeq™3000/4000和HiSeq™X采用的是RTA2。在MiSeq或者HiSeq™系统测平衡性低的文库时,建议根据平台和控制软件版本的不同至少参入5-10% phix。
MiniSeq,NextSeq500/550和NovaSeq 6000平台采用的是两通道化学。采用这种技术,每个cycle中会根据两种染料以及两张图片来确定参入的碱基。这使得测序的速度有所提高,数据处理也更高效。NextSeq 500/550和MiniSeq采用RTA2进行图片处理,碱基读取以及质量值的计算。NovaSeq 6000采用的RTA3已经做过优化,可以节省数据处理时间。对于采用两色化学的平台,碱基平衡性对于color matrix correction以及光强校正尤为重要。对于平衡性低的文库,在MiniSeq或者NextSeq测序,建议最低参入10%的phix,在NovaSeq测序建议最低参入5% phix。
iSeq 100平台是illumina唯一采用单通道化学的测序平台。每个测序Cycle采用单一的染料,两张图片,两次化学步骤(在每个拍照步骤前分别有一个化学步骤)。 iSeq100采用RTA2来进行光强读取,碱基读取以及质量值计算。在iSeq™100上测碱基平衡性低的文库,建议至少加入5%的phix。
相关资源:
For any feedback or questions regarding this article (Illumina Knowledge Article #7132), contact Illumina Technical Support [email protected].
Last updated
Was this helpful?
