使用BaseSpace基因云计算平台和bcl2fastq2 2.17以及以上版本软件时,如何排查拆分异常情况
本技术文档适用于BaseSpace基因云计算平台和bcl2fastq2 2.17以及以上版本软件的数据拆分问题。
BaseSpace基因云计算平台和bcl2fastq2 v2.17+在数据拆分结束后均会产生拆分总结文件,即DemuxSummaryF1L#.txt。其中,L#代表的是流动槽(Flow Cell)上的通道(Lane)编号,并且针对每条Lane都会产生一个对应的文件。该文件可以用于排查拆分异常情况的原因。
在哪里可以看到DemuxSummaryF1L#.txt
bcl2fastq2 2.17以及以上版本软件
FASAQ文件生成结束后,在stats文件夹中会产生DemuxSummaryF1L#.txt文件。Stats文件夹位于指定的输出目录中。
BaseSpace基因云计算平台
针对所有的测序运行runs,在FASAQ文件生成结束后,系统会把DemuxSummaryF1L#.txt存储在相应的project中。用户可以通过点击project中分析中的FASTQ Generation链接,在弹出的Summary界面点击View Files(图1),就可以在文件列表中找到DemuxSummaryF1L#.txt文件。
进入 Run 页面,在 “Latest Analyses” 下选择 FASTQ Generation 链接(见图 1)。

图 1:列出 FASTQ Generation 分析的 Run Summary 页面
2. 在 Analysis Info 页面中,向下滚动到 Log Files(见图 2)。你可以在日志文件列表中找到 DemuxSummaryF1L#.txt 文件。

图 2:Analysis 页面中的日志文件列表
DemuxSummaryF1L#.txt含有哪些内容?
DemuxSummaryF1L#.txt含有两个部分。正如图3显示的,文件的上半部分是一个tab分割的表格,该表格总结了每个tile的样本拆分情况。表格的左侧是流动槽上的tile列表。 在表格的顶部,样品按输入样品表的顺序列出。 样品0指的是未拆分的reads。 下表显示每个tiles上reads拆分到每个样本的百分比。 通常,在所有tiles上针对某一个样本的拆分比例应该是接近的。用户可以利用tile的总结信息明确与特定tile有关的拆分问题。
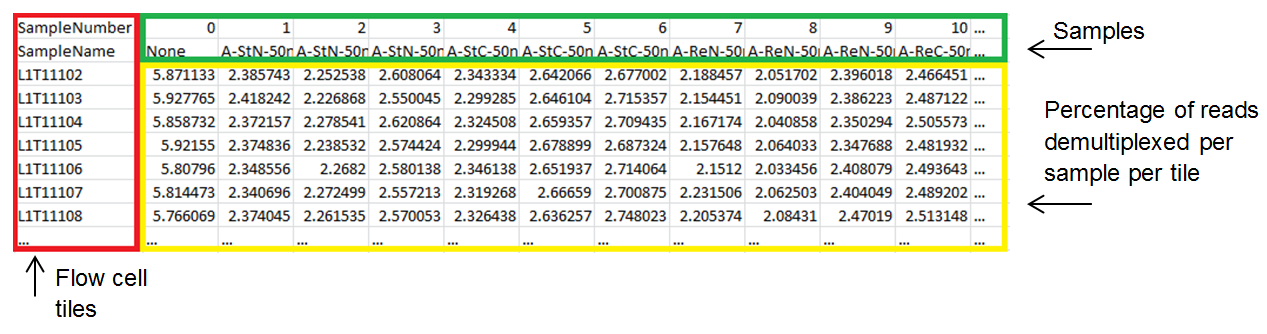
图 3:Tile summary 部分显示了按 tile 统计拆分到各个样本的 reads 百分比。
DemuxSummaryF1L#.txt文件(图4)的第二部分列出了前1000个比较多的未拆分的index序列以及对应的数目(或者说分配到每个index序列的cluster的数目)

图4:DemuxSummaryF1L#.txt文件中index序列部分截图,该部分列出了前1000个没有拆分到样本的index序列
当遇到拆分问题时,可以使用此列表将预期index序列(SampleSheet中)和测序测到的实际序列进行比较。
这些列表可以揭示一些低拆分比例的常见原因:
在样本表(SampleSheet.csv)输入了错误方向的index序列
在样本表中输入了错误的index(比如Nextera vs TruSeq UD或者index A001 vs index A006)
不同lane的样本发生了混合
For any feedback or questions regarding this article (Illumina Knowledge Article #7134), contact Illumina Technical Support [email protected].
Last updated
Was this helpful?
