Infinium基因分型芯片manifest文件中Chr或MapInfo列的值为0时代表什么意思?
为什么有些markers会比对到0号染色体上?
图1. Manifest文件中Chr=0位点示例
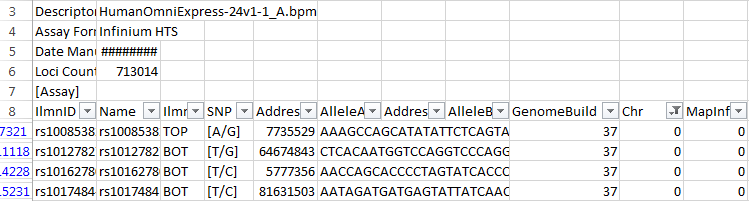
在制作manifest文件时,首先探针序列会被比对到manifest文件GenomeBuild列中指定版本的参考基因组上,然后SNP或者indel的坐标信息就会被记录在MapInfo这一列中。在manifest文件中,如果一个marker的Chr或MapInfo记录为0,那么有以下两种可能的解释:
该探针没有有效的比对结果
该探针有超过1个以上最优得分的比对结果
我们如何知道为什么一个探针的Chr信息标注为0?
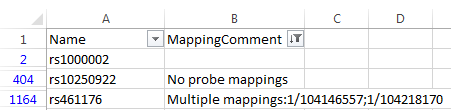
针对在manifest文件中Chr/MapInfo为0的位点,请参考Illumina.com官网产品支持文件中的比对备注(Mapping Comments)文件。这个文件的Mapping Comments列提供了为什么这些位点标注为0:
Blank – 说明一个位点的探针序列没有复杂的比对信息。在manifest文件中该位点的Chr和MapInfo会标注为这个最优比对位置信息。
No probe mappings – 说明一个位点的探针序列比对到指定版本的参考基因组上没有有效的比对结果。在manifest文件中该位点的Chr和MapInfo会标注为0。
Multiple mappings – 说明一个位点的探针序列有多个最优得分的比对结果,这些最优的比对位置信息会以分号为分隔符罗列在Multiple mappings后面。在manifest文件中Chr和MapInfo会标注为0。
图2. 备注文件比对举例
其他资源
如果想了解更多有关芯片的manifest文件的信息,请参考以下的技术公告
Infinium Genotyping Manifest Column Headings .
For any feedback or questions regarding this article (Illumina Knowledge Article #7313), contact Illumina Technical Support [email protected].
Last updated
Was this helpful?
