FASTQ文件解读
Illumina测序技术使用 簇生成和边合成边测序(SBS)化学技术对流动槽(flow cell)上数百万或数十亿簇(cluster)进行测序,具体簇的数目取决于测序平台。 在边合成边测序化学过程中,仪器上的实时分析(RTA)软件对每个簇的每个循环进行碱基检出和存储。 RTA以单个读取碱基(base call,或称BCL)文件的形式存储碱基检出数据。 测序完成后,必须将BCL文件中的测定的碱基转换为序列数据。 此过程称为BCL到FASTQ的转换。
FASTQ文件是一个文本文件,其中包含通过流动槽(flow cell)上质控参数的簇(cluster)的测序数据(有关簇的质控参数,请参阅本公告的“其他信息”部分)。如果样本是multiplexed,则FASTQ文件生成的第一步是demultiplexing。 demultiplexing根据簇的index序列将簇分配给样本。 demultiplexing后,将每个样本的组合序列写入FASTQ文件。 如果未对样品进行multiplex,则不会发生demultiplexing,并且对于每个流动槽每个通道(Lane)中的所有簇都分配给一个样品。
对于单端测序的运行,将为每个流动槽上每条通道的每个样品创建一个Read 1(R1)FASTQ文件。 对于双端测序的运行,将为每个流动槽上每条通道的每个样品各创建一个R1和一个Read 2(R2)FASTQ文件。 FASTQ文件是使用扩展名*.fastq.gz压缩和创建的。
FASTQ文件是什么样的?
对于每个通过质控参数的簇,一个序列被写入相应样本的R1 FASTQ文件,而对于双端测序运行,另外一个序列也被写入该样本的R2 FASTQ文件。 FASTQ文件中的每个条目包含4行:
序列标识符,其中包含有关测序运行和簇的信息。 该行的具体内容会因使用的BCL到FASTQ转换软件而不同。
序列(碱基信号; A,C,T,G和N)。
分隔符,只是一个加号(+)。
这是R1 FASTQ文件中单个记录条目的示例:
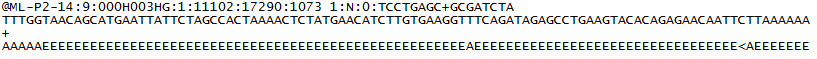
如何查看FASTQ文件
FASTQ文件最多可以包含数百万个条目,大小可以为数兆字节或千兆字节,这常常使它们太大而无法在常规文本编辑器中打开。 通常情况下,并没有必要查看FASTQ文件,因为它们是做下游分析(例如与参考基因组序列比对或从头组装)的中间文件。
如果出于故障排除目的或兴趣需要查看FASTQ文件时,则需要在可以处理非常大文件的文本编辑器打开文件,或者使用可以通过命令行查看大文件的Unix或Linux系统。
如何生成FASTQ文件
FASTQ文件生成是MiSeq上MiSeq Reporter和MiniSeq上的Local Run Manager进行所有分析工作流程的第一步。 分析完成后,FASTQ文件位于MiSeq上的< run folder > \ Data \ Intensities \ BaseCalls和MiniSeq上的< run folder > \ Alignment _#\ <子文件夹> \ Fastq中。
对于上传到BaseSpace基因云计算平台的所有运行,测序数据上传结束后会自动生成FASTQ文件,并且FASTQ文件可以用作BaseSpace基因云计算平台上各种分析apps的输入文件。 在BaseSpace基因云计算平台上,您可以在与您的运行关联的项目(projects)中找到FASTQ文件。
bcl2fastq转换软件可用于将目前所有Illumina测序系统上产生的数据转换成FASTQ文件。
有关在FASTQ文件生成过程中使用的不同设置的详细信息,请参阅下面的软件用户指南。
其他信息
有关簇通过质控参数的说明和要求,请参阅MiSeq: Imaging and Base Calling 课程的1.5.8节。
有关NovaSeq,NextSeq 500/550和MiniSeq系统上的碱基检出的更多信息,请参阅2-Channel SBS Technology。
有关在MiSeq和HiSeq系统上进行碱基检出的更多信息,请参见Illumina Sequencing Technology。
For any feedback or questions regarding this article (Illumina Knowledge Article #7412), contact Illumina Technical Support [email protected].
Last updated
Was this helpful?
